新型コロナウイルス(COVID-19)陽性患者数のグラフ 2
前回、新型コロナウイルスの患者のグラフをオープンデータから抜き出してグラフにしました。それだけだと、本当に意味がない感じだったので、近似式をだして予測してみました。
累計のグラフの出し方です。3月1日からのグラフにしています。
sr = df.cumsum()
fig = plt.figure(figsize=(12, 6))
ax = fig.add_subplot(111, xlabel="Date", ylabel="累計")
fig.suptitle('東京都 陽性患者数 累計', fontsize=20)
ax.bar(sr['2020-03-01':].index,sr['2020-03-01':].values)
ax.grid(True)
plt.xticks(rotation=30)
fig.savefig('corona2.png',bbox_inches="tight")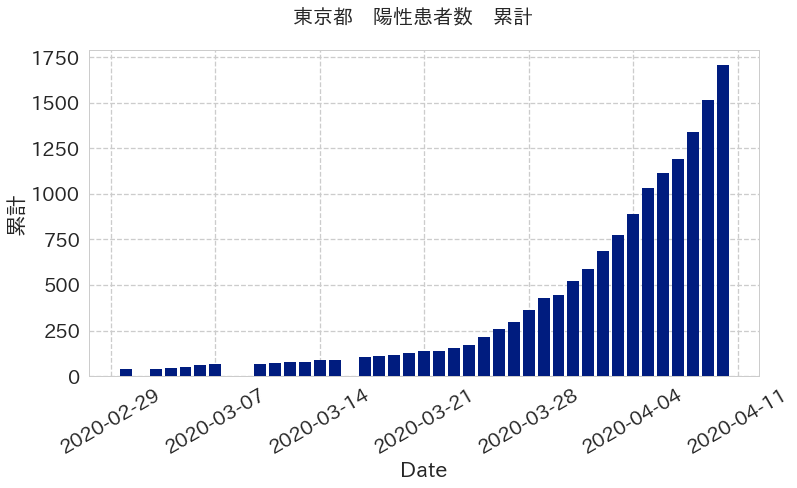
累乗近似をしてみました。
#NaNとなっているデータに0を入れるsr = sr.asfreq('D') sr = sr.fillna(0)#curve_fitをつかうfrom scipy.optimize import curve_fit import numpy as npdef nonliner_fit(x, a, b, c): return bx*a + c x = np.array(range(len(sr))) y = sr.values p_opt,cov = curve_fit(nonliner_fit,x,y) p_opt y_fit = np.vectorize(nonliner_fit)
グラフにしてみます。
fig = plt.figure(figsize=(12,8),dpi=100)
ax = fig.add_subplot(1,1,1)
fig.suptitle('東京都 陽性患者数 累計', fontsize=20)
ax.plot(x,y,lw=0,marker='o',clip_on=False,)
X=np.array(range(len(sr)))
ax.plot(X,y_fit(X,p_opt[0],p_opt[1],p_opt[2]),lw=2,color="red")
peri=-(-(len(sr))//7)
x1 = pd.date_range(sr.index[1],periods=peri, freq='7d')
xyobi = []
for i in range(peri):
a=1+i*7
xyobi.append(a)
plt.xticks(xyobi,x1.strftime("%y-%m-%d"))
plt.xticks(rotation=60)
ax.legend(["Value", "Approximation"])
plt.show()
fig.savefig('corona01.png',bbox_inches="tight")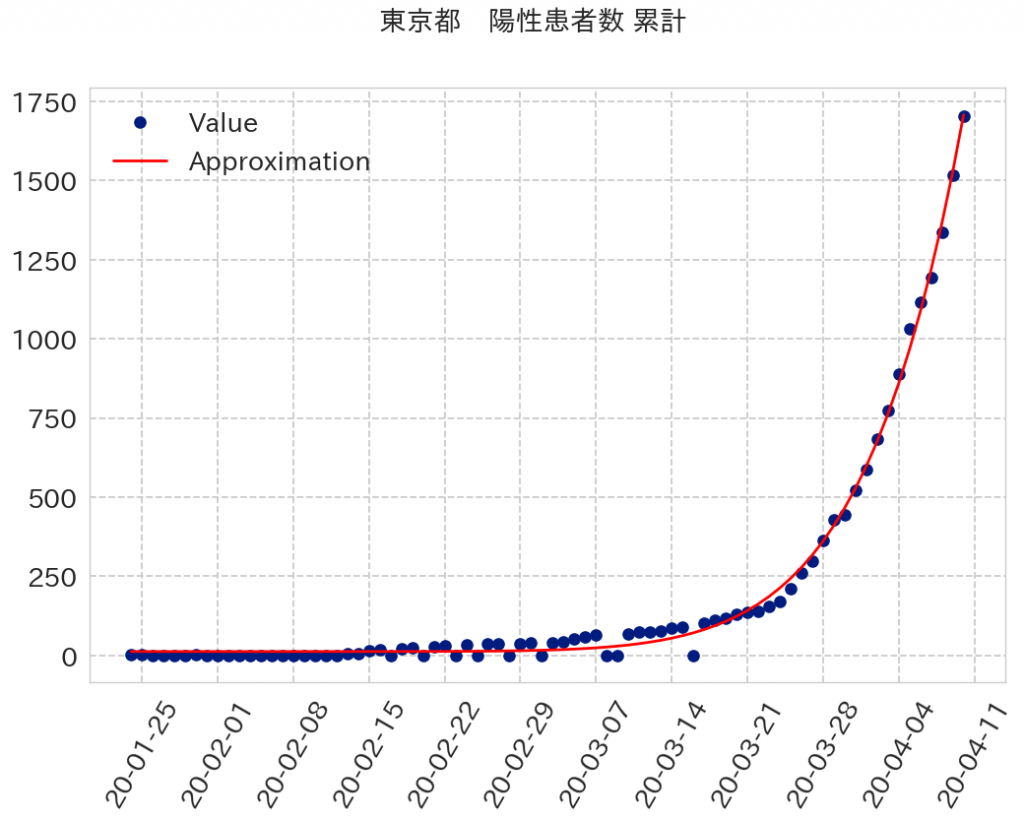
いい感じじゃないですか。実際、こんなスピードで感染者が増えちゃうと困っちゃうんで、なんとか緩やかになってほしいですね。ちなみに、10日後、30日後、60日後、90日後までこの近似式で予測してみました。
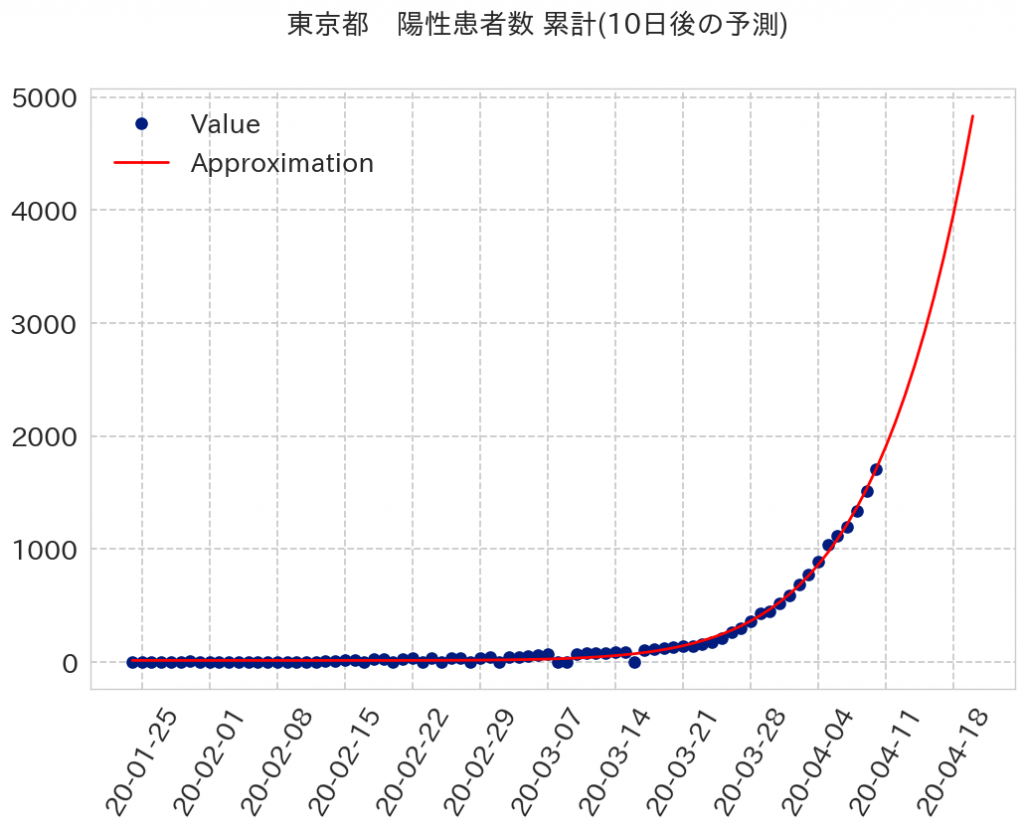
10日後には5000人近くまで感染者が増えますね。
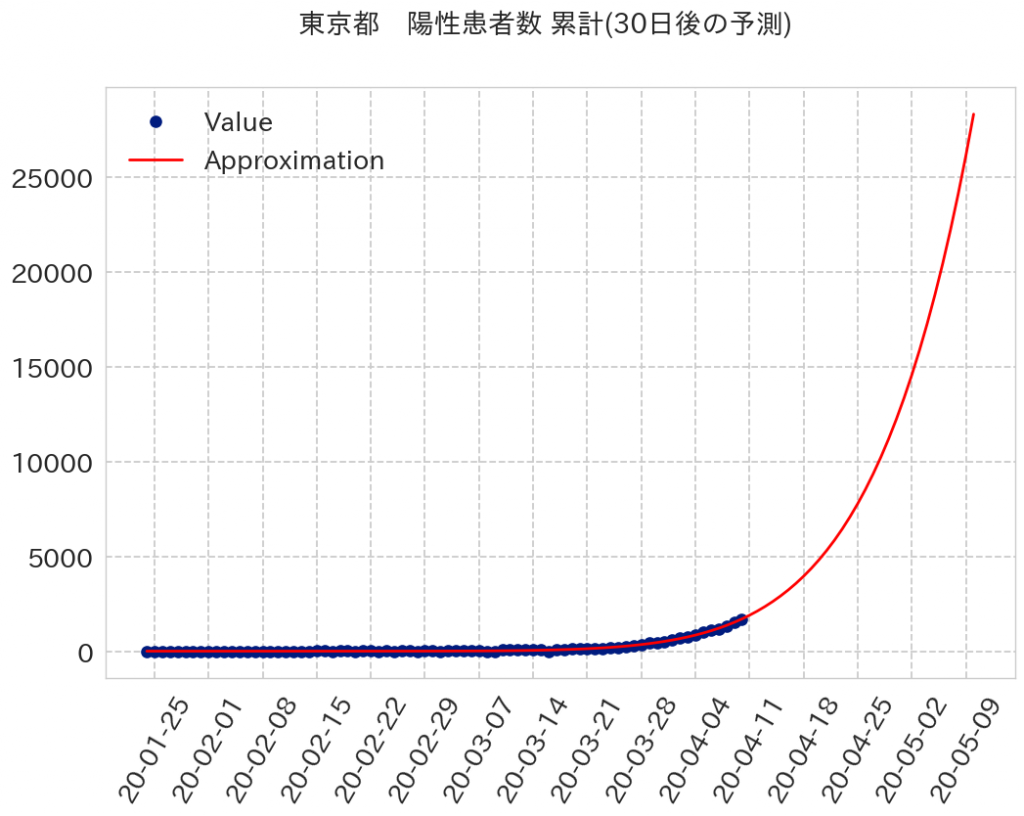
30日後には2万8千人近くまで感染者が増えますね。
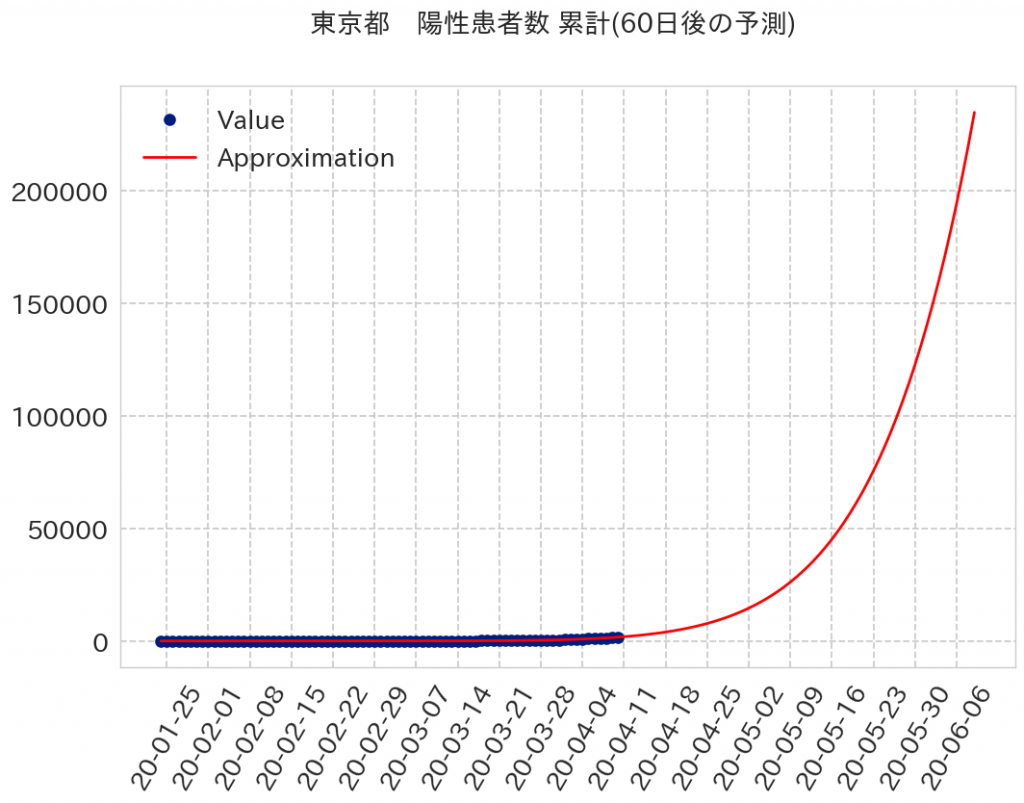
60日後の梅雨に入るときには23万人超えですね。現時点では1750人程度なので爆発的に増えている感じになりますね。こんなになってたら梅雨と相まって相当な憂鬱になりそうですね。
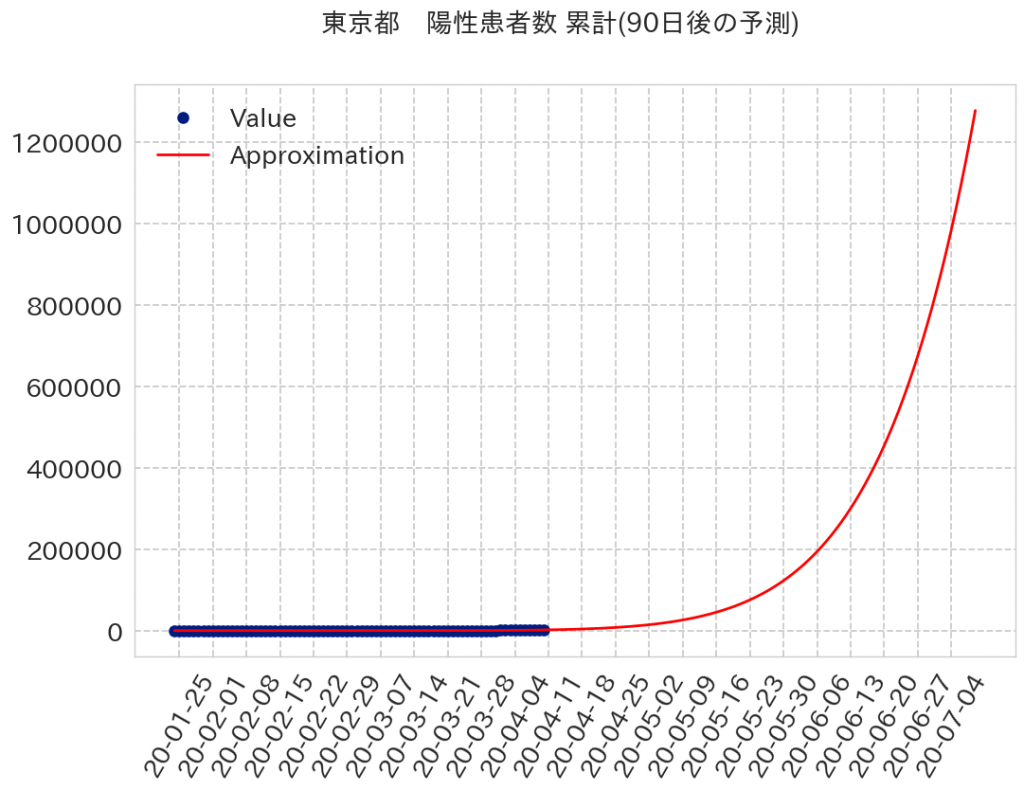
7月で120万人を超えちゃいますね。ここまできたら猛暑とコロナで地獄ですね。致死率が3.5%で計算すると4万2千人が死んでしまうことになります。結構な数ですね。そうならないために今、外出は控えたほうが良いですね。
以上
関連する投稿:
- 2020-04-19:新型コロナウイルス(COVID-19)陽性患者数の予測と結果
- 2020-04-11:新型コロナウイルス(covid-19)陽性患者数のグラフ 1
- 2014-09-04:Pyhton Apacheアクセスログを取り込んでグラフで表示する
- 2016-11-19:マンション契約率とマンション販売戸数のグラフ化
- 2017-04-21:以前予測したPVの結果はどうであったか
“新型コロナウイルス(COVID-19)陽性患者数のグラフ 2” への1件の返信